|
|||
タンパク質超二次構造コードディープラーニング解析
ディープラーニング予測システムSSSCPredsを用いたSARS-CoV-2多重変異株の柔軟性/剛性マップパターン及びその定量化データが、感染性及び中和回避能の良い指標となることに関する論文が公表されました(https://doi.org/10.1021/acsomega.1c03055)。SSSCPredsは以下のサイトから自由にダウンロード可能です(https://staff.aist.go.jp/izumi.h/SSSCPreds/index-e.html)。
AI技術を以下のタンパク質超二次構造コードと組み合わせてアミノ酸配列のみからタンパク質のコンフォメーション可変性を予測するプログラムを開発しました。構造未知だったSARS-CoV-2タンパク質を用いてコンフォメーションの予測精度の検証を進めています(https://doi.org/10.1021/acsomega.0c04472)。
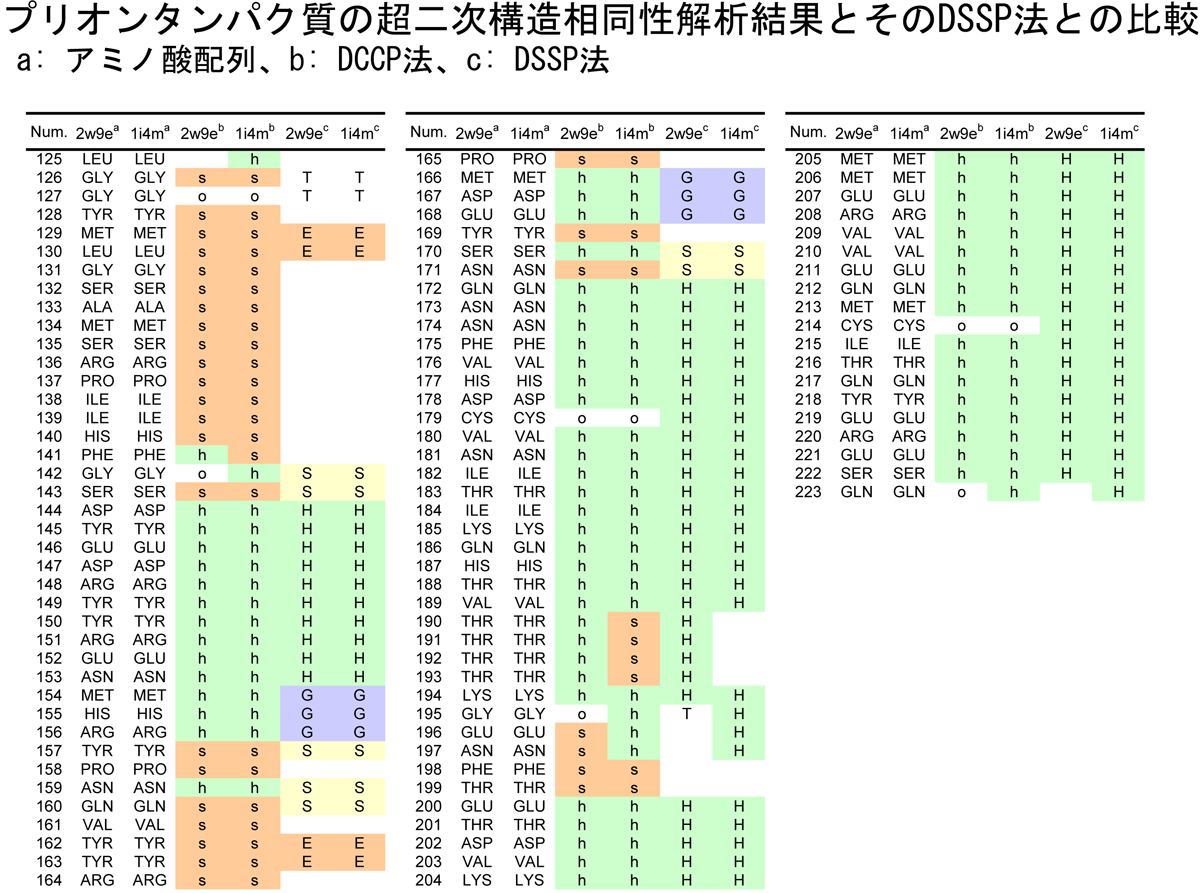
タンパク質の二次構造の帰属にDSSP法(Kabsch, W.; Sander, C. Biopolymers 1983, 22, 2577-2637)が標準的手法として広く用いられています。しかしながら、DSSP法はコード化手法としてはloopもしくはirregularに対応する、符号化されないアミノ酸残基のブランクがかなりの頻度で存在するため、データマイニングの用途には適していませんでした。
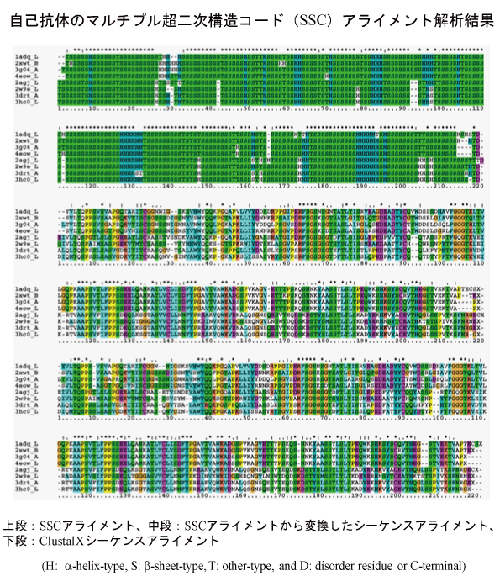
当研究グループで開発したDCCP法(Dictionary of Conformational Code in Proteins)は、タンパク質X線結晶構造データの3D情報を1D情報に変換することで、そのコンフォメーションをαへリックス型パターン h、βシート型パターン s、その他の型のパターン o の3種類の符号のみで記述することができます。特に、複雑な構造を持ったloopのような超二次構造のデータマイニングに有効で、構造は似ていながらアミノ酸配列が大きく異なるタンパク質の多型解析に役立ちます。
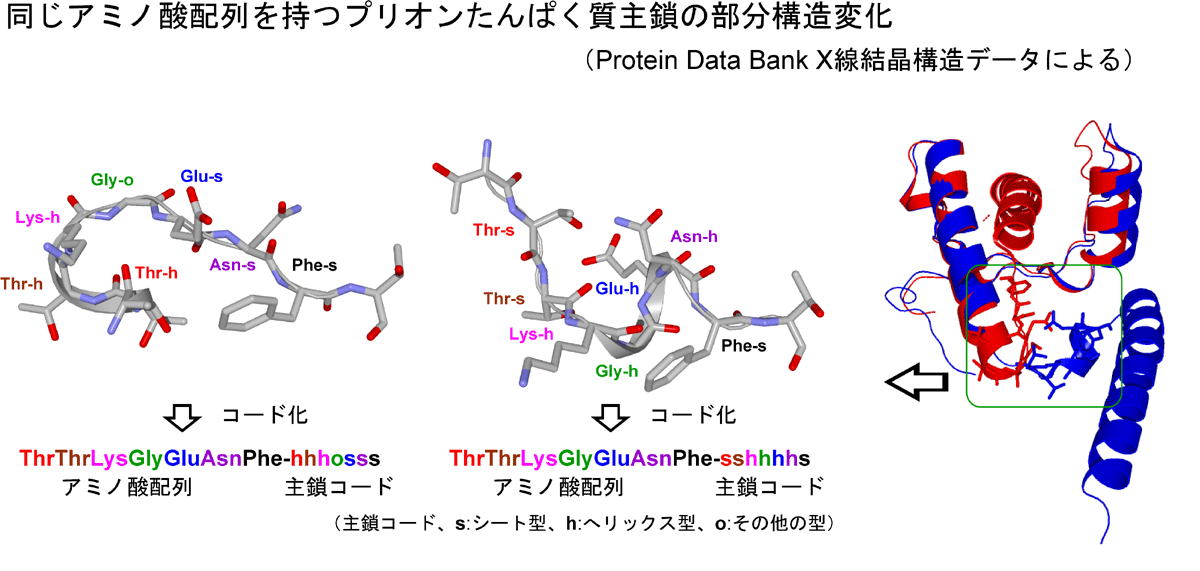
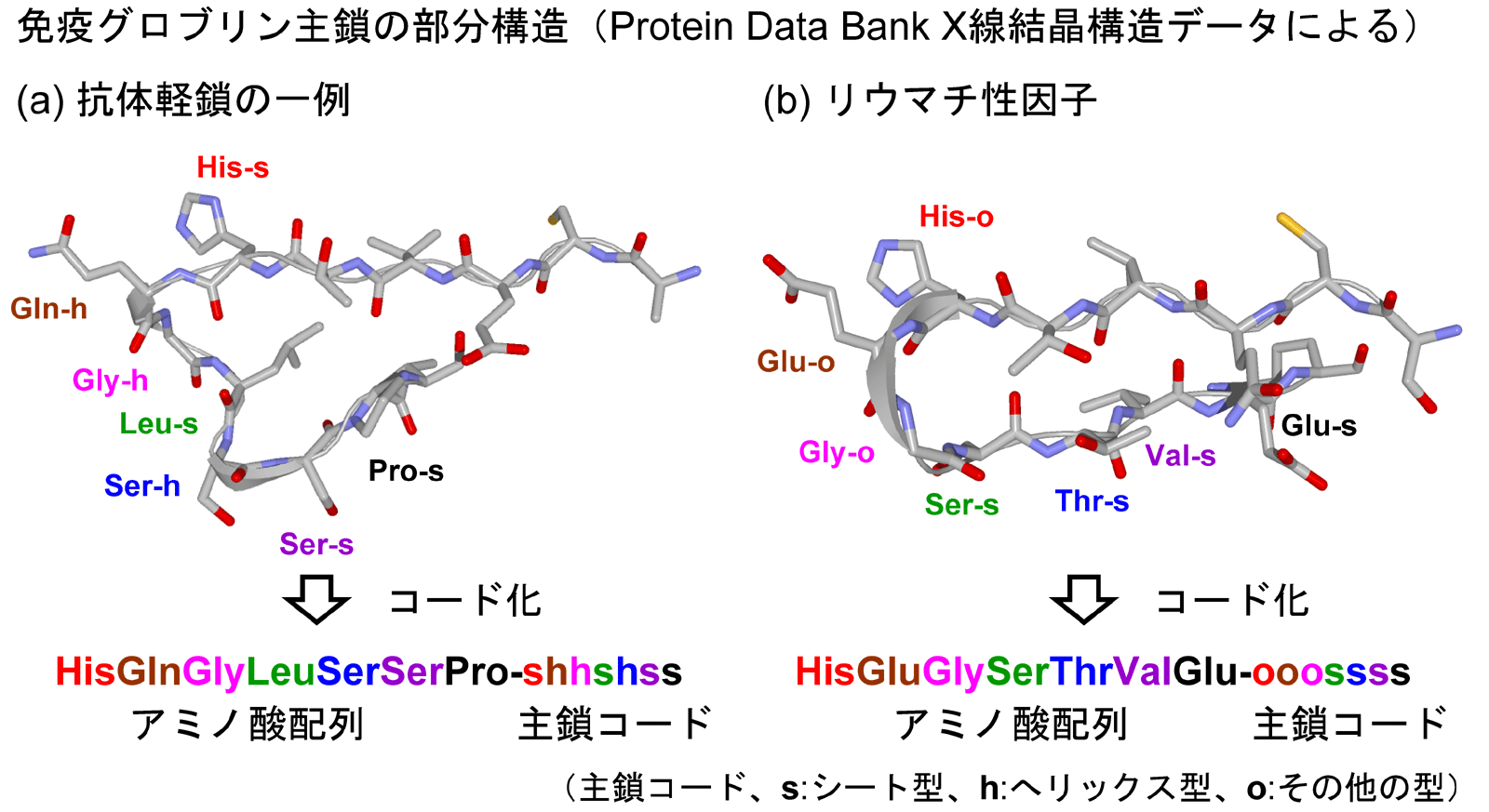
本超二次構造相同性解析手法を用いることで、世界ではじめて免疫グロブリンとMHC分子に共通する特徴的なフラグメント構造(shhshss)を持つことを見出しました(J. Chem. Inf. Model., DOI: 10.1021/ci300420d)。興味深いことに、リュウマチ性因子をはじめとする自己抗体はこのフラグメント構造を持っていません。また、MHCクラスI分子のβ-2マイクログロブリンが持つこのフラグメント構造は構造を保持したまま、正常細胞に対する免疫細胞活性化の閾値を 上げて攻撃を抑制する機能を持つLILR分子と相互作用することが見出されています。
このように、タンパク質のコンフォメーションをコード化してデータマイニングする手法は、既存のバイオインフォマティクス手法と組み合わせることにより、システムバイオロジーのメタデータとしての活用が期待できます。
さらに、汎用性を高めるため、ClustalXをはじめとするマルチプルアライメント解析ソフトウェアにそのまま適用可能な超二次構造コード(SSSC)を開発しており、タンパク質PDBデータから直接自動変換可能です。
マルチプルアライメント解析用SSSC変換プログラムについては、無償のフリーウェアとして配布しておりますので、ご興味のある方はお問い合わせください。
また、タンパク質のコード化変換データは無償で配布可能ですので、ご興味のある方はお問い合わせください。その構造パターン解析法については知財化(特願2010-116862、PCT/JP2008/051673(特願2007-77133) )を進めておりますので、ソフトウェア化に興味を持つ企業関係者の方のご連絡をお待ちしております。
タンパク質超二次構造コード相同性解析プログラム(SSSC Analysis)
本研究の一部は、科研費(JP19K05431)の助成を受けて行われたものです。