亀田 倫史のホームページ
(暫定版:随時更新中)
Sorry, this page is japanese only (under construction)

主に、分子動力学(MD)シミュレーションを用いて、蛋白質・核酸・薬物(化合物)・カーボン素材(カーボンナノチューブなど)を研究しています。
人工知能やバイオインフォマティクスを利用して遺伝子配列を再設計する研究も行っています。
最近では、液液相分離(liquid-liquid phase separation)の研究も始めています。
ポスドク募集中!興味がある方ご連絡ください。
連絡先:kameda-tomoshi AT aist.go.jp
もともと私は、蛋白質が変性した構造がどのように天然構造に巻き戻るか、研究を行ってきました。(蛋白質巻き戻り過程、protein folding prathway)
手法としては、1アミノ酸を少数の球で表現した粗視化モデルや(Proteins 53 616-28 (2003))、
AMBER、CHARMMなどの全原子モデルを用いたシミュレーションを用いてきました。(Proc. Natl. Acad. Sci. 103 (47) 17765-17770 (2006))
そのころに培った技術をもとに、現在では多数のテーマについて研究を行っており、多くは実験家と共同で研究を行っています。
非常に少ない実験データを学習データとして用いても、高機能な蛋白質を設計できることがわかってきました。
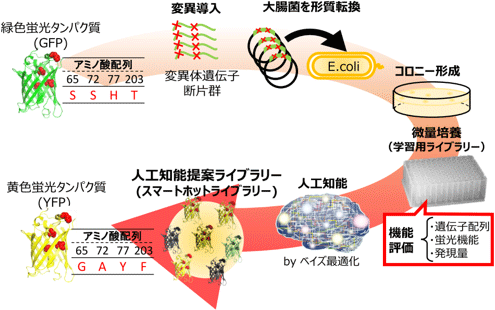
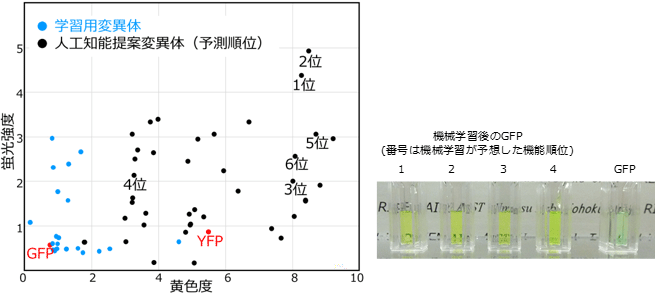
プレスリリース
https://www.aist.go.jp/aist_j/press_release/pr2018/pr20180831/pr20180831.html
Yutaka Saito, Misaki Oikawa, Hikaru
Nakazawa, Teppei Niide, Tomoshi Kameda, Koji
Tsuda, Mitsuo Umetsu
ACS Synth. Biol. 2018, 7, 9, 2014–2022
2


プレスリリース
https://www.aist.go.jp/aist_j/press_release/pr2019/pr20190606/pr20190606.html
Yutaka Saito, Wataru Kitagawa, Toshitaka Kumagai, Naoyuki Tajima, Yoshiyuki Nishimiya, Koichi Tamano, Yoshiaki Yasutake, Tomohiro Tamura, Tomoshi Kameda
Sci Rep. 2019; 9: 8338
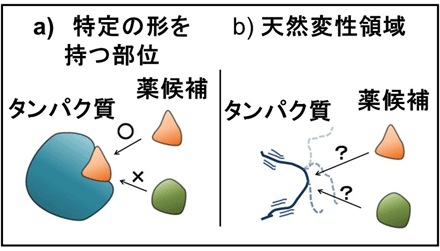
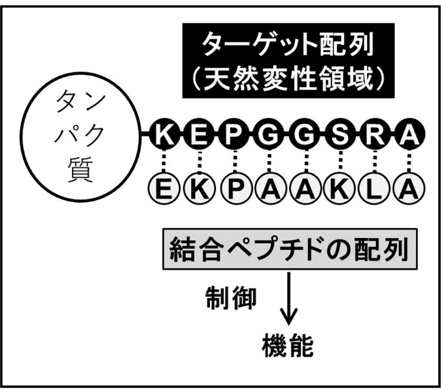
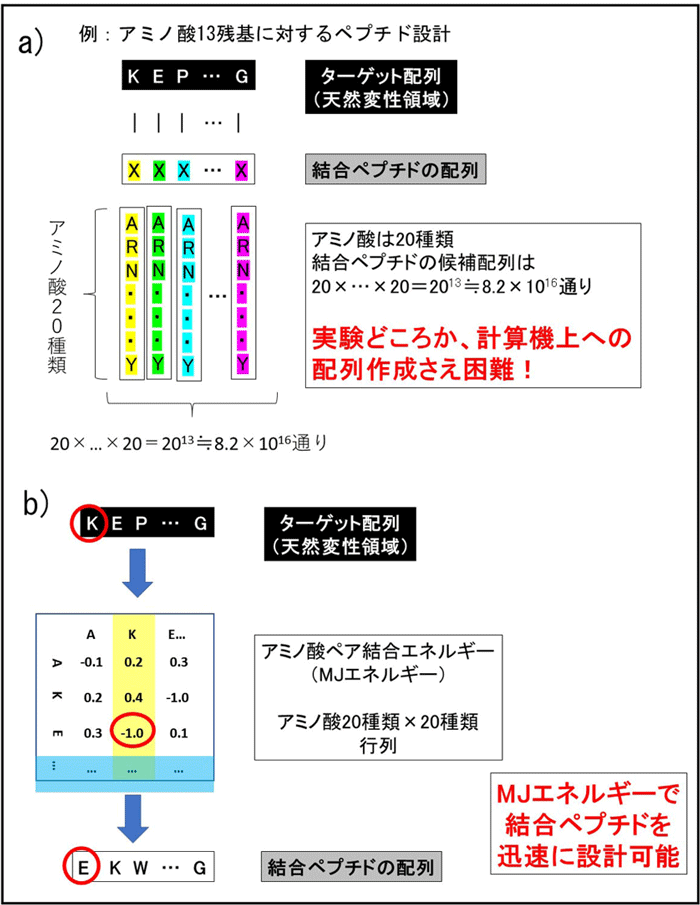
3)天然変性領域を標的としたペプチドの人工設計
現在、蛋白質に結合する化合物・ペプチドを予想・設計する研究は、それらの立体構造に基づいて行われています。
しかし、もともと構造を形成しない「天然変性領域」に対する分子設計は困難です。
我々は、そのような領域に対しても、結合力が強いペプチドを簡単に設計できる手法を考案し、
実際に天然変性領域を持つ蛋白質p53で、実際に結合することを実験によって確認しました。
プレスリリース
https://www.aist.go.jp/aist_j/press_release/pr2019/pr20190628/pr20190628.html
"Rational design using sequence information only produces a peptide that binds to the intrinsically disordered region of p53"
Sci. Rep. 9, Article number: 8584 (2019)
4
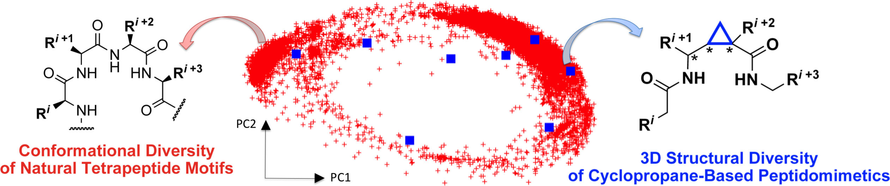
, , Kuwahara, Hideyuki, Endoh, , , , Yamano, , Arisawa, and Shuto
“Cyclopropane-Based Peptidomimetics Mimicking Wide-Ranging Secondary Structures of Peptides: Conformational Analysis and Their Use in Rational Ligand Optimization”
Chemistry a Europian Journal (2017) (23) 13, 3159–3168
5
我々は
また、
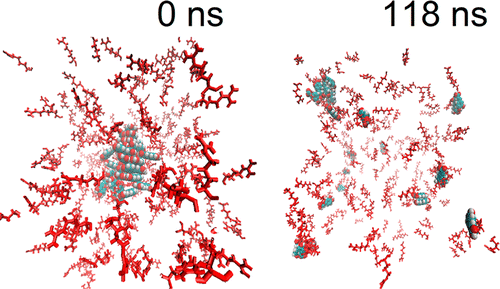
Hirano, Atsushi#; Kameda, Tomoshi#; Shinozaki, Daisuke; Arakawa, Tsutomu; Shiraki, Kentaro,
"Molecular Dynamics Simulation of the Arginine-Assisted Solubilization of Caffeic Acid: Intervention in the Interaction"
J Phys Chem. b 117 (25), 7518–7527 (2013) #: equally
contributed
6
抗体医薬は非常に効果が高い薬物の一つで、その市場規模は10兆円を超えようとしています。一方で、価格が非常に高額であることが問題となっています。その原因の一つは抗体をカラム精製する際に、酸性溶液を用いるため抗体が失活し、収率が下がることです。
アルギニンはカラム溶出にも効果を発揮します。
電荷が正、負、電荷なしのカラム担体を用いて、溶出の実験を行ったところ、3つとも効果を持ち、その効果は担体の電荷に依らないことを示しました。特に、電荷が正、負の場合については、pH7下での抗体溶出の実験を行い、実際に効果があることを確認しています。また、理論計算により、アルギニンは、様々な相互作用によって担体と結合することから、その効果を発揮することを示しました。
(例:CaptoMMCを担体とするカラムと様々な溶液を用いて抗体を溶出し、Native PAGEしたもの。食塩、エチレングリコールと比較し、アルギニン水溶液を用いると溶出できることがわかる。)
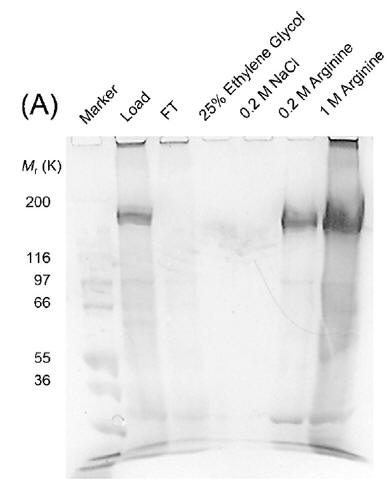
Atsushi Hirano; Tsutomu Arakawa; TOMOSHI Kameda*
"Interaction of Arginine with Capto MMC in Multimodal Chromatography"
Journal of Chromatography A, 1338:58-66 (2014)
7
カーボンナノチューブ(CNT)は、強度が高い、熱・電気伝導率が高いなどの特異な性質を持ち、様々な分野で用いられています。薬学ではDDS(ドラッグデリバリー)に用いるなどの応用研究がされています。
しかし、CNTはベンゼン環のお化けのような形をしており、水溶性が非常に悪く、その応用を困難なものとしています。
ポリフェノール類と同様、アルギニンでの分散を試みたところ、難溶性が高いためか、効果はありませんでした。そこで、ポリアルギニン、ポリリジンを試したところ、分散させることに成功し、ポリアルギニンのほうが効果が高く、また長いほうがより効果があることがわかりました。
CNTとペプチドを混ぜたMD計算を行うと、アルギニンは側鎖のグアニジウム基がCNTに強く張り付くことで効果を発揮することがわかりました。
他にも、蛋白質とCNTの相互作用、CNTによるペプチド・アミノ酸の酸化還元制御(システインやグルタチオン)などの研究を行っています。
例:ポリアルギニン、ポリリジンによるCNTの分散

Atsushi Hirano, Takeshi Tanaka, Hiromichi Kataura, and Tomoshi
Kameda*
“Arginine Side Chains as a Dispersant for Individual Single-Wall Carbon Nanotubes”
Chemistry a Europian Journal, 20(17):4922-30
(2014)
8)MD計算に基づく、RNA熱安定性の推定
MDシミュレーションの結果は正確なのか、その確からしさについて長い間議論が行われてきました。
2010年代に入り、DavidShawらが、MD計算しかできけれども、その計算速度は世界一というAntonという計算機を開発しました。これを用いて、変性構造から「かなり」長時間MD計算をすると、12蛋白質のうち、10蛋白質が正しい立体構造に巻き戻ること(Science 2011)、蛋白質・薬物複合体から薬物をはがして離れた位置に置き、こちらも「かなり」長時間MD計算をすると、正しい位置に巻き戻る(JACS 2012)ことを示しました。
つまり、MDシミュレーションは、多くの場合において「かなり」長時間計算を行えば、正しい結果を再現することを証明しました。(彼らはAntonを用いて1msのMD計算を行いましたが、これは2017現在世界記録です(Nature, 2010))
我々は、核酸、特にRNAでもMD計算は正しいのか調べようと思いました。しかし、Antonは非常に高価なマシンで、手に入れることは難しく(〇億円らしい)、彼らのように長時間計算することはできません。
そこで、自由エネルギー摂動法(FEP)とレプリカ交換MD法を組み合わせた手法を用いて、「短くはないけれど、そんなに長くない」計算時間で、9つのRNA二重らせんの熱安定性(ΔG)を計算したところ、実験結果との相関係数がR2=0.97、実験値との誤差が~0.55kcal/molと、非常に良い結果を示しました。これは、MDシミュレーション用いれば、実験とほぼ同等の結果を得ることができることを意味します。
これは、東京大・桜庭俊さんとの共同研究です。
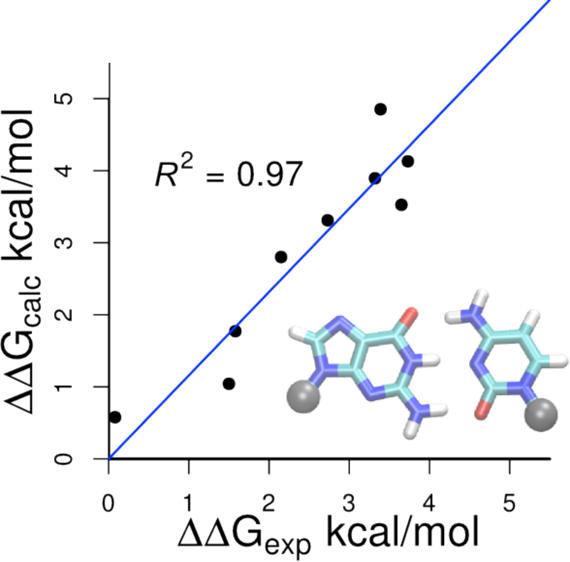
Sakuraba, Shun; Asai, Kiyoshi; Kameda, Tomoshi*
“Predicting RNA Duplex Dimerization Free-energy Changes Upon Mutations Using Molecular Dynamics Simulations”
J Phys Chem Lett (2015) Nov 5;6(21):4348-51
9)dockingによる、蛋白質・RNA複合体立体構造の予測
蛋白質と薬物候補分子とのdocking計算(複合体立体構造予測)は、様々なソフトウェアがあり、広く用いられてきましたが、核酸を扱うことができるソフトは、ほとんどない状況が続いてきました。
先ほど述べたように、MDシミュレーションの精度は、RNAについても正確であることがわかりました。そこで、このMDで用いられている分子に関する情報(電荷・vdW力の強度など。力場(force field)という)を、dockingシミュレーション用ソフトzdockに移植し、蛋白質・RNA複合体予測を行ったところ、世界最高水準の精度で予測することに成功しました。
これは東京大・岩切さんとの共同研究です。
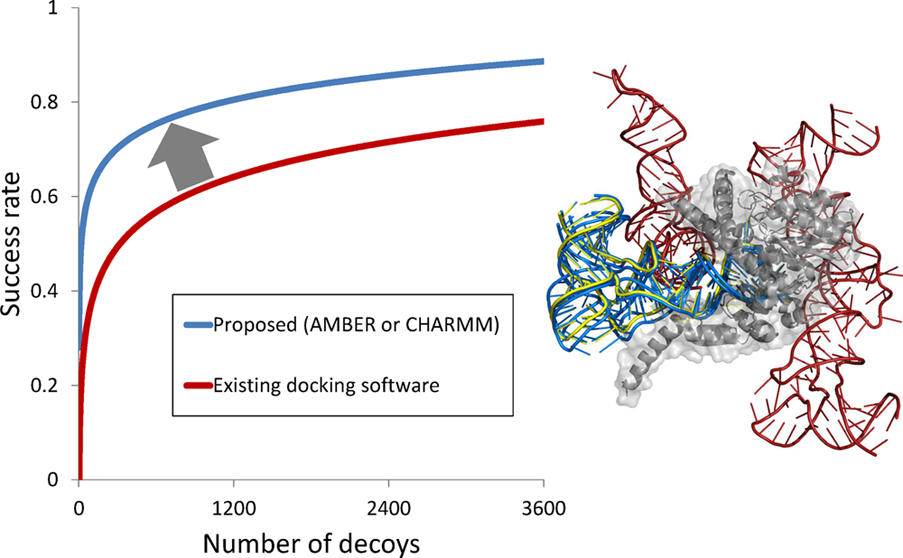
Junichi Iwakiri, Michiaki Hamada, Kiyoshi Asai, and Tomoshi Kameda*
“Improved accuracy in RNA-protein rigid body docking by incorporating force field for molecular dynamics simulation into the scoring function”
J. Chem. Theory Comput., (2016) 12(9):4688-97
10
<学歴>
1987(昭和62)年3月
青森市立浦町小学校卒業
1990(平成2)年3月 青森市立浦町中学校卒業
1990(平成2)年4月 青森県立青森高等学校入学
1993(平成5)年3月 青森県立青森高等学校卒業
1994(平成6)年4月 京都大学理学部理学科入学
1998(平成10)年3月 京都大学理学部理学科卒業(主に物理を修める)
1998(平成10)年4月京都大学大学院理学研究科化学専攻修士課程入学(郷信宏研究室)
2000(平成12)年3月京都大学大学院理学研究科化学専攻修士課程修了
2000(平成12)年4月 神戸大学大学院自然科学研究科分子集合科学専攻博士課程入学(高田彰ニ研究室)
2004(平成16)年3月 神戸大学大学院自然科学研究科分子集合科学専攻博士課程修了
2004(平成16)年3月博士号(理学)取得(神戸大学)
<職歴>
2004(平成16)年4月 - 2007年9月 産業技術総合研究所 産総研特別研究員
2007(平成19)年10月-2013年2月 産業技術総合研究所 研究員
2013(平成25)年3月-2022年3月 産業技術総合研究所 主任研究員
2022(令和4)年4月から 産業技術総合研究所 上級主任研究員
2011(平成22)年5月から2023年3月まで 北海道大学大学院 生命科学院 客員准教授
email:
kameda-tomoshiATMARKaist.go.jp
(ATMARKを@に直してください)
<論文(国際誌)>
1. Atsushi Hirano, Momoyo Wada, Takehiro K. Sato, Tomoshi Kameda, N-acetyl amino acid amide solubility in aqueous 1,6-hexanediol solutions: Insights into the protein droplet deformation mechanism: International Journal of Biological Macromolecules (2024)
2. Atsushi Hirano, Tomoshi Kameda, Momoyo Wada, Takeshi Tanaka, Hiromichi Kataura, Solubilization of Carbon Nanobelts in Aqueous Solutions: Optical and Colloidal Properties: Nano Letters (2023)
3. Motomi Matsuda, Keisuke Ikeda, Tomoshi Kameda, Hiroyuki Nakao, Minoru Nakano, Fine-Tuning and Enhancement of pH-Dependent Membrane Permeation of Cyclic Peptides by Utilizing Noncanonical Amino Acids with Extended Side Chains.: Journal of medicinal chemistry (2023)
4. Shinji Iida, Tomoshi Kameda, Dissociation Rate Calculation via Constant-Force Steered Molecular Dynamics Simulation.: Journal of chemical information and modeling (2023)
5. Atsushi Hirano, Tomoshi Kameda, Momoyo Wada, Takeshi Tanaka, Hiromichi Kataura, Coenzyme corona formation on carbon nanotubes leads to disruption of the redox balance in metabolic reactions.: Nanoscale 15(5) 2340-2353 (2023)
6. Ahmed El-Refaey, Daichi Kozawa, Tomoshi Kameda, Yuichiro K. Kato, Yoshihiro Ito, Masuki Kawamoto, Diameter-Selective Sorting of Single-Walled Carbon Nanotubes Using π-Molecular Tweezers for Energy Materials: ACS Applied Nano Materials 6(3) 1919-1926 (2023)
7. Tomoyuki Ito, Thuy Duong Nguyen, Yutaka Saito, Yoichi Kurumida, Hikaru Nakazawa, Sakiya Kawada, Hafumi Nishi, Koji Tsuda, Tomoshi Kameda, Mitsuo Umetsu, Selection of target-binding proteins from the information of weakly enriched phage display libraries by deep sequencing and machine learning.: mAbs 15(1) 2168470-2168470 (2023)
8. Shinji Iida, Kameda Tomoshi, Free energy and kinetic rate calculation via non-equilibrium molecular simulation: application to biomolecules: Biophysical Reviews 14(6) 1303-1314 (2022)
9. Kiyoto Kamagata, Maulana Ariefai, Hiroto Takahashi, Atsumi Hando, Dwiky Rendra Graha Subekti, Keisuke Ikeda, Atsushi Hirano, Tomoshi Kameda, Rational peptide design for regulating liquid-liquid phase separation on the basis of residue-residue contact energy.: Scientific reports 12(1) 13718-13718 (2022)
10. Tomoto Ura, Tomoshi Kameda, Fina A Laksmi, Matsujiro Ishibashi, Tsutomu Arakawa, Kentaro Shiraki, Atsushi Hirano, Affinity of phenolic compounds for transition metal ions immobilized on cation-exchange columns.: Journal of chromatography. A 1676 463277-463277 (2022)
11. Tomohito Nakayama, Kaito Kobayashi, Tomoshi Kameda, Muneaki Hase, Atsushi Hirano, Protein’s Protein Corona: Nanoscale Size Evolution of Human Immunoglobulin G Aggregates Induced by Serum Albumin: ACS Applied Materials & Interfaces 14(29) 32937-32947 (2022)
12. Moe Yamazaki, Keisuke Ikeda, Tomoshi Kameda, Hiroyuki Nakao, Minoru Nakano, Kinetic Mechanism of Amyloid-β-(16–22) Peptide Fibrillation: The Journal of Physical Chemistry Letters 13(26) 6031-6036 (2022)
13. Yosuke Iizumi, Yoshihiro Sowa, Wakana Goi, Yuichi Aono, Motoki Watanabe, Yoichi Kurumida, Tomoshi Kameda, Kenichi Akaji, Masatoshi Kitagawa, Toshiyuki Sakai, Stabilization of CDK6 by ribosomal protein uS7, a target protein of the natural product fucoxanthinol.: Communications biology 5(1) 564-564 (2022)
14. Motoki Watanabe, Shogen Boku, Kaito Kobayashi, Yoichi Kurumida, Mamiko Sukeno, Mitsuharu Masuda, Katsura Mizushima, Chikage Kato, Yosuke Iizumi, Kiichi Hirota, Yuji Naito, Michihiro Mutoh, Tomoshi Kameda, Toshiyuki Sakai, A chemoproteoinformatics approach demonstrates that aspirin increases sensitivity to MEK inhibition by directly binding to RPS5: PNAS Nexus 1(2) pgac059 (2022)
15. Yuki Terauchi, Megumi Nagayama, Takumi Tanaka, Hiroki Tanabe, Akira Yoshimi, Kei Nanatani, Hiroshi Yabu, Toshihiko Arita, Takeshi Higuchi, Tomoshi Kameda, Keietsu Abe, Adsorption Kinetics and Self-Assembled Structures of Aspergillus oryzae Hydrophobin RolA on Hydrophobic and Charged Solid Surfaces: APPLIED AND ENVIRONMENTAL MICROBIOLOGY 88(6) (2022)
16. Ryo Kitahara, Tomoshi Kameda, Phase separation by biopolymers: Basics and applications.: Biophysics and physicobiology 19 e190028 (2022)
17. Mizuki Watanabe, Makoto Nagata, Ryohei Doi, Mai Uemura, Nanase Ochiai, Wataru Ichinose, Koichi Fujiwara, Yoshihiro Sato, Tomoshi Kameda, Koh Takeuchi, Satoshi Shuto, Helix-forming aliphatic homo-δ-peptide foldamers based on conformational restriction effects of cyclopropane: Organic & Biomolecular Chemistry 21(5) 970-980 (2022)
18. Yutaka Saito, Misaki Oikawa, Takumi Sato, Hikaru Nakazawa, Tomoyuki Ito, Tomoshi Kameda, Koji Tsuda, Mitsuo Umetsu, Machine-Learning-Guided Library Design Cycle for Directed Evolution of Enzymes: The Effects of Training Data Composition on Sequence Space Exploration: ACS CATALYSIS 11(23) 14615-14624 (2021)
19. Tomoyuki Ito, Hafumi Nishi, Tomoshi Kameda, Mayu Yoshida, Reito Fukazawa, Sakiya Kawada, Hikaru Nakazawa, Mitsuo Umetsu, Combination Informatic and Experimental Approach for Selecting Scaffold Proteins for Development as Antibody Mimetics: CHEMISTRY LETTERS 50(11) 1867-1871 (2021)
20. Jinzen Ikebe, Munenori Suzuki, Aya Komori, Kaito Kobayashi, Tomoshi Kameda, Enzyme modification using mutation site prediction method for enhancing the regioselectivity of substrate reaction sites: SCIENTIFIC REPORTS 11(1) 8059-8059 (2021)
21. Shujie Li, Takuya Yoshizawa, Ryota Yamazaki, Ayano Fujiwara, Tomoshi Kameda, Ryo Kitahara, Pressure and Temperature Phase Diagram for Liquid-Liquid Phase Separation of the RNA-Binding Protein Fused in Sarcoma: JOURNAL OF PHYSICAL CHEMISTRY B 125(25) 6821-6829 (2021)
22. Naoyuki Tajima, Toshitaka Kumagai, Yutaka Saito, Tomoshi Kameda, Comparative analysis of the relationship between translation efficiency and sequence features of endogenous proteins in multiple organisms: Genomics 113(4) 2675-2682 (2021)
23. Atsushi Hirano, Momoyo Wada, Takehiro K. Sato, Tomoshi Kameda, The solubility of N-acetyl amino acid amides in organic acid and alcohol solutions: Mechanistic insight into structural protein solubilization: INTERNATIONAL JOURNAL OF BIOLOGICAL MACROMOLECULES 178 607-615 (2021)
24. Shin Irumagawa, Kaito Kobayashi, Yutaka Saito, Takeshi Miyata, Mitsuo Umetsu, Tomoshi Kameda, Ryoichi Arai, Rational thermostabilisation of four-helix bundle dimeric de novo proteins.: Scientific reports 11(1) 7526-7526 (2021)
25. Kiyoto Kamagata, Rika Chiba, Ichiro Kawahata, Nanako Iwaki, Saori Kanbayashi, Kana Maeda, Hiroto Takahashi, Atsushi Hirano, Koji Fukunaga, Keisuke Ikeda, Tomoshi Kameda, Characterization of design grammar of peptides for regulating liquid droplets and aggregates of FUS.: Scientific reports 11(1) 6643-6643 (2021)
26. Atsushi Hirano, Tomoshi Kameda, Aromaphilicity Index of Amino Acids: Molecular Dynamics Simulations of the Protein Binding Affinity for Carbon Nanomaterials: ACS APPLIED NANO MATERIALS 4(3) 2486-2495 (2021)
27. Motoki Watanabe, Shogen Boku, Kaito Kobayashi, Yoichi Kurumida, Mitsuharu Masuda, Chikage Kato, Mamiko Sukeno, Katsura Mizushima, Yosuke Iizumi, Kiichi Hirota, Yuji Naito, Michihiro Mutoh, Tomoshi Kameda, and Toshiyuki Sakai, A chemoproteoinformatics approach demonstrates that aspirin increases sensitivity to MEK inhibition by directly binding to RPS5. PNAS Nexus (2021)
28. Yutaka Saito, Misaki Oikawa, Takumi Sato, Hikaru Nakazawa, Tomoyuki Ito, Tomoshi Kameda, Koji Tsuda, Mitsuo Umetsu, Machine-Learning-Guided Library Design Cycle for Directed Evolution of Enzymes: The Effects of Training Data Composition on Sequence Space Exploration, ACS Catal. 11(23) 14615-14624 (2021).
29. Atsushi Hirano, Tomoshi Kameda, Aromaphilicity Index of Amino Acids: Molecular Dynamics Simulations of the Protein Binding Affinity for Carbon Nanomaterials. ACS Appl. Nano Mater. 4(3) 2486-2495 (2021).
30. Tomoyuki Ito, Hafumi Nishi, Tomoshi Kameda, Mayu Yoshida, Reito Fukazawa, Sakiya Kawada, Hikaru Nakazawa, Mitsuo Umetsu “Combination Informatic and Experimental Approach for Selecting Scaffold Proteins for Development as Antibody Mimetics” Chemistry Letter 50, 11, 1867-1871(2021) (IF=1.4)
31. Jinzen Ikebe, Munenori Suzuki, Aya Komori, Kaito Kobayashi, Tomoshi Kameda “Enzyme modification using mutation site prediction method for enhancing the regioselectivity of substrate reaction sites” Scientific Reports 11, 19004 (2021)
32. Yutaka Saito, Misaki Oikawa, Takumi Sato, Hikaru Nakazawa, Tomoyuki Ito, Tomoshi Kameda, Koji Tsuda, Mitsuo Umetsu “Machine-Learning-Guided Library Design Cycle for Directed Evolution of Enzymes: The Effects of Training Data Composition on Sequence Space Exploration” ACS Catal. 11, 23, 14615–14624 (2021) (IF=13.1)
33. Shujie Li, Takuya Yoshizawa, Ryota Yamazaki, Ayano Fujiwara, Tomoshi Kameda, Ryo Kitahara “Pressure and Temperature Phase Diagram for Liquid–Liquid Phase Separation of the RNA-Binding Protein Fused in Sarcoma” J. Phys. Chem. B 125, 25, 6821–6829 (2021) (IF=3.0)
34. Naoyuki Tajima, Toshitaka Kumagai, Yutaka Saito, Tomoshi Kameda “Comparative analysis of the relationship between translation efficiency and sequence features of endogenous proteins in multiple organisms” Genomics 113, 4, 2675-2682 (2021) (IF=5.7)
35. Atsushi Hirano, Momoyo Wada, Takehiro K Sato, Tomoshi Kameda “The solubility of N-acetyl amino acid amides in organic acid and alcohol solutions: Mechanistic insight into structural protein solubilization” International Journal of Biological Macromolecules 178, 607-615 (2021) (IF=7.0)
36. Shin Irumagawa, Kaito Kobayashi, Yutaka Saito, Takeshi Miyata, Mitsuo Umetsu, Tomoshi Kameda, Ryoichi Arai “Rational thermostabilisation of four-helix bundle dimeric de novo proteins” Scientific Reports 11, 7526 (2021)
37. Kiyoto Kamagata, Rika Chiba, Ichiro Kawahata, Nanako Iwaki, Saori Kanbayashi, Kana Maeda, Hiroto Takahashi, Atsushi Hirano, Koji Fukunaga, Keisuke Ikeda, Tomoshi Kameda “Design grammar characterization of peptides for regulating liquid droplets and aggregates of FUS” Scientific Reports 11, 6643(2021)
38. Atsushi Hirano, Tomoshi Kameda “Aromaphilicity Index of Amino Acids: Molecular Dynamics Simulations of the Protein Binding Affinity for Carbon Nanomaterials” ACS Applied Nano Materials 4(3) 2486-2495(2021)
39. 10. A Hirano, M Wada, TK Sato, T Kameda “The solubility of N-acetyl amino acid amides in organic acid and alcohol solutions: Mechanistic insight into structural protein solubilization” International Journal of Biological Macromolecules 178, 607-615 (2021)
40. Motoki Watanabe, Yasumasa Yamada, Yoichi Kurumida, Tomoshi Kameda, Mamiko Sukeno, Mahiro Iizuka-Ohashi, Yoshihiro Sowa, Yosuke Iizumi, Hideki Takakura, Shingo Miyamoto, Toshiyuki Sakai, Michihiro Mutoh “Rabdosianone I, a Bitter Diterpene from an Oriental Herb, Suppresses Thymidylate Synthase Expression by Directly Binding to ANT2 and PHB2” Cancers 13(5):982 (2021)
41. Yoichi Kurumida, Yutaka Saito & Tomoshi Kameda “Predicting antibody affinity changes upon mutations by combining multiple predictors” Scientific Reports 10 Article number: 19533 (2020)
42. T Wakamoto, S Kitazawa, T Kameda, R Kitahara “Dynamic aspects of pressure and temperature‐stabilized intermediates of outer surface protein A” Proteins: Structure, Function, and Bioinformatics 88, 11, 1423-1433 (2020)
43. Shujie Li, Huan Yao, Tomoshi Kameda, Wei Jiang, Ryo Kitahara “Volumetric Properties for the Binding of 1, 4-Dioxane to Amide Naphthotubes in Water” J Phys Chem b 124, 41, 9175-9181 (2020)
44. Shun Sakuraba, Junichi Iwakiri, Michiaki Hamada, Tomoshi Kameda, Genichiro Tsuji, Yasuaki Kimura, Hiroshi Abe, Kiyoshi Asai “Free-energy calculation of ribonucleic inosines and its application to nearest-neighbor parameters” J Chem Theory Comp 16, 9, 5923-5935(2020)
45. Kiyoto Kamagata, Saori Kanbayashi, Masaya Honda, Yuji Itoh, Hiroto Takahashi, Tomoshi Kameda, Fumi Nagatsugi & Satoshi Takahashi “Liquid-like droplet formation by tumor suppressor p53 induced by multivalent electrostatic interactions between two disordered domains” Scientific Reports 10, 580 (2020)
46. Suguru Nishinami, Tomoshi Kameda, Tsutomu Arakawa, Kentaro Shiraki ” Hydantoin and Its Derivatives Reduce the Viscosity of Concentrated Antibody Formulations by Inhibiting Associations via Hydrophobic Amino Acid Residues” Ind. Eng. Chem. Res. 58, 36, 16296-16306 (2019)
47. Kiyoto Kamagata, Eriko Mano, Yuji Itoh, Takuro Wakamoto, Ryo Kitahara, Saori Kanbayashi, Hiroto Takahashi, Agato Murata, Tomoshi Kameda” Rational design using sequence information only produces a peptide that binds to the intrinsically disordered region of p53” Scientific Reports 9, Article number: 8584 (2019)
48. Atsushi Hirano, Kazuki Iwashita, Tomoto Ura, Shun Sakuraba, Kentaro Shiraki, Tsutomu Arakawa, Tomoshi Kameda “The binding affinity of uncharged aromatic solutes for negatively charged resins is enhanced by cations via cation–π interactions: The case of sodium ion and arginine” Journal of Chromatography A 1595, 97-107 (2019)
49. Yutaka Saito, Wataru Kitagawa, Toshitaka Kumagai, Naoyuki Tajima, Yoshiyuki Nishimiya, Koichi Tamano, Yoshiaki Yasutake, Tomohiro Tamura, Tomoshi Kameda “Developing a codon optimization method for improved expression of recombinant proteins in actinobacteria” Scientific Reports 9, Article number: 8338 (2019)
50. Tomohiko Matsuzawa, Masahiro Watanabe, Tomoshi Kameda, Akihiko Kameyama, Katsuro Yaoi “Cooperation between β‐galactosidase and an isoprimeverose‐producing oligoxyloglucan hydrolase is key for xyloglucan degradation in Aspergillus oryzae” FEBS J 286, 16, 3182-3193 (2019)
51. Yuko Shishido, Fumiaki Tomoike, Keiko Kuwata, Haruka Fujikawa, Yoshitaka Sekido, Yuko Murakami‐Tonami, Tomoshi Kameda, Naoko Abe, Yasuaki Kimura, Satoshi Shuto, Hiroshi Abe A “Covalent Inhibitor for Glutathione S‐Transferase Pi (GSTP1‐1) in Human Cells.” Chem Bio Chem 20, 9 900-905 (2019) IF=2.8
52. Yutaka Saito, Misaki Oikawa, Hikaru Nakazawa, Teppei Niide, Tomoshi Kameda, Koji Tsuda*, and Mitsuo Umetsu*. “Machine-Learning-Guided Mutagenesis for Directed Evolution of Fluorescent Proteins.” ACS Synthetic Bio. 7 (9), pp 2014–2022 (2018) IF=5.3
53. Ryo Kitahara*, Shun Sakuraba, Tomoshi Kameda, Sanshiro Okuda, Mengjun Xue and Frans A.A. Mulder. “Nuclear magnetic resonance-based determination of dioxygen binding sites in protein cavities.” Protein Science 27, 769-779 (2018) IF=2.5
54. Atsushi Hirano, Tomoshi Kameda, Momoyo Wada, Takeshi Tanaka, Hiromichi Kataura. “Carbon Nanotubes Facilitate Oxidation of Cysteine Residues of Proteins.” J. Phys. Chem. Lett. 8, 5216-5221 (2017). IF=9.3
55. Kobayashi Y1, Kameda T, Hoshino M, Fujii N, Ohno H, Oishi S., “Fe(II)-Complexation of tripodal hexapeptide ligands with three bidentate triazolylpyridines: induction of metal-centred chirality by peptide macrocyclization.” Dalton Trans. 2017 Oct 17;46(40):13673-13676. IF=4.0
56. Yuko Shishido, Fumiaki Tomoike, Yasuaki Kimura, Keiko Kuwata, Takato Yano, Kenji Fukui, Haruka Fujikawa, Yoshitaka Sekido, Yuko Murakami-Tonami, Tomoshi Kameda, Satoshi Shuto, Hiroshi Abe “A covalent G-site inhibitor for glutathione S-transferase Pi (GSTP1-1)” Chem. Commun., 2017, Advance Article IF=6.3(from 2015)
57. Takaaki Kurinomaru, Kengo Kuwada, Shunsuke Tomita, Tomoshi Kameda, Kentaro Shiraki “Noncovalent PEGylation through Protein–Polyelectrolyte Interaction: Kinetic Experiment and Molecular Dynamics Simulation” J. Phys. Chem. B, 2017, 121 (28), pp 6785–6791IF=3.2(from 2015)
58. Satoshi Takano, Takayoshi Tsuzuki, Takashi Murayama, Tomoshi Kameda, Yasuhiro Kumaki, Takashi Sakurai, Hayato Fukuda, Mizuki Watanabe, Mitsuhiro Arisawa, Satoshi Shuto “Synthesis of 8-Substituted Analogues of Cyclic ADP-4-Thioribose and Their Unexpected Identification as Ca2+-Mobilizing Full Agonists”J. Med. Chem., 2017, 60 (13), pp 5868–5875
59. Atsushi Hirano, Tomoshi Kameda, Shun Sakuraba, Momoyo Wada, Takeshi Tanaka, Hiromichi Kataura “Disulfide Bond Formation of Thiols by Carbon Nanotubes” Nanoscale, 2017, 9 (17), 5389-5393, IF=7.4 (from 2014)
60. Takahiro Kawamura, Takuro Wakamoto, Soichiro Kitazawa, Shun Sakuraba, Tomoshi Kameda, and Ryo Kitahara “Analysis of O2-binding sites in proteins using gas-pressure NMR spectroscopy: outer surface protein A” Biophys J , 2017, 112, 9, 1820-1828 IF=4.0 (from 2014)
61. Yoshiaki Yasutake, Tomoshi Kameda and Tomohiro Tamura “Structural insights into the mechanism of the drastic changes in the enzymatic activity of cytochrome P450 vitamin D3 hydroxylase (CYP107BR1) caused by a mutation distant from the active site” Acta Cryst. (2017). F73, 266-275 IF=0.65(from 2014)
62. Akira Mizuno, Tomoshi Kameda, Tomoki Kuwahara, Hideyuki, Endoh, Yoshihiko Ito, Shizuo Yamada, Kimiko Hasegawa, Akihito Yamano, Mizuki Watanabe, Mitsuhiro Arisawa, and Satoshi Shuto “Cyclopropane-Based Peptidomimetics Mimicking Wide-Ranging Secondary Structures of Peptides: Conformational Analysis and Their Use in Rational Ligand Optimization”Chemistry a Europian Journal (2017) (23) 13, 3159–3168 IF=5.7 (from 2014)
63. Atsushi Hirano, Takuya Maruyama, Kentaro Shiraki, Tsutomu Arakawa, Tomoshi Kameda* “A study of the small-molecule system used to investigate the effect of arginine on antibody elution in hydrophobic charge-induction chromatography”Protein Expression and Purification, (2016) (16) 30215-7 IF=1.7(from 2014)
64. Junichi Iwakiri, Michiaki Hamada, Kiyoshi Asai, and Tomoshi Kameda* “Improved accuracy in RNA-protein rigid body docking by incorporating force field for molecular dynamics simulation into the scoring function” J. Chem. Theory Comput., (2016) 12(9):4688-97 IF=5.5(from2014)
65. Atsushi Hirano, Tomoshi Kameda, Yohei Yomogida, Momoyo Wada, Takeshi Tanaka, Hiromichi Kataura “Origin of the Surfactant-Dependent Redox Chemistry of Single-Wall Carbon Nanotubes” Chem Nano Mat (2016) 2; 9, 911-920
66. Takatoshi Sato, Mizuki Watanabe, Takayoshi Tsuzuki, Satoshi Takano, Takashi Murayama, Takashi Sakurai, Tomoshi Kameda, Hayato Fukuda, Mitsuhiro Arisawa, and Satoshi Shuto “Design, Synthesis, and Identification of 4″α-Azidoethyl-cyclic ADP-Carbocyclic-ribose as a Highly Potent Analogue of Cyclic ADP-Ribose, a Ca2+-Mobilizing Second Messenger” J. Med. Chem., (2016) 59 (15), pp 7282–7286; IF=5.4(from 2014)
67. Kitahara R, Yoshimura Y, Xue M, Kameda T, Mulder FA. “Detecting O2 binding sites in protein cavities.” Sci Rep. (2016) Feb 2; 6: 20534. IF=5.6(from 2014)
68. Tamaki H, Egawa A, Kido K, Kameda T, Kamiya M, Kikukawa T, Aizawa T, Fujiwara T, Demura M. “Structure determination of uniformly (13)C, (15)N labeled protein using qualitative distance restraints from MAS solid-state (13)C-NMR observed paramagnetic relaxation enhancement.” J Biomol NMR. (2016) Jan;64(1):87-101. IF=3.1(from 2014)
69. Sakuraba, Shun; Asai, Kiyoshi; Kameda, Tomoshi* “Predicting RNA Duplex Dimerization Free-energy Changes Upon Mutations Using Molecular Dynamics Simulations” J Phys Chem Lett (2015) Nov 5;6(21):4348-51. IF=7.5(from 2014)
70. Goro Terai, Junichi Iwakiri, Tomoshi Kameda, Michiaki Hamada and Kiyoshi Asai “Comprehensive prediction of lncRNA–RNA interactions in human Transcriptome” BMC Genomics (2016) Jan 11;17 Suppl 1:12. IF=4.0(from 2014)
71. Hirano A, Arakawa T, Kameda T*. “Effects of arginine on multimodal anion exchange chromatography”Protein Expression and Purification, (2015) Dec;116:105-12. IF=1.7(from 2014)
72. Zha Li, Tomoshi Kameda, Takashi Isoshima, Eiry Kobatake, Takeshi Tanaka, Yoshihiro Ito, and Masuki Kawamoto “Solubilization of Single-Walled Carbon Nanotubes using a Peptide Aptamer in Water below Critical Micelle Concentration” Langmuir, 31 (11), pp 3482–3488(2015) IF=4.5(from 2014)
73. Takaaki Kurinomaru, Tomoshi Kameda, and Kentaro Shiraki “Effects of Multivalency and Hydrophobicity of Polyamines on Enzyme Hyperactivation of a-Chymotrypsin” Journal of Molecular Catalysis B: Enzymatic, 115:135-139 (2015) IF=2.1(from 2014)
74. Takayoshi Tsuzuki, Satoshi Takano, Natsumi Sakaguchi, Takashi Kudoh, Takashi Murayama,Takashi Sakurai, Minako Hashii, Haruhiro Higashida, KarinWeber, Andreas H. Guse, Tomoshi Kameda, Takatsugu Hirokawa, Yasuhiro Kumaki, Mitsuhiro Arisawa, Barry V. L. Potter, and Satoshi Shuto “Design, Synthesis, and Chemical and Biological Properties of Cyclic ADP-4-Thioribose as a Stable Equivalent of Cyclic ADP-Ribose” MESSENGER Vol. 3, 35–51, (2014)
75. Hirano A, Maruyama T, Shiraki K, Arakawa T, Kameda T*. “Mechanism of protein desorption from 4-mercaptoethylpyridine resins by arginine solutions” Journal of Chromatography A, 1373:141-8. (2014). IF=4.2
76. Atsushi Hirano; Tsutomu Arakawa; TOMOSHI Kameda* “Interaction of Arginine with Capto MMC in Multimodal Chromatography”Journal of Chromatography A, 1338:58-66 (2014) IF=4.2
77. Atsushi Hirano, Takeshi Tanaka, Hiromichi Kataura, and Tomoshi Kameda*“Arginine Side Chains as a Dispersant for Individual Single-Wall Carbon Nanotubes” Chemistry a Europian Journal, 20(17):4922-30 (2014) IF=5.7
78. Soichiro Kitazawa, Tomoshi Kameda, Ayumi Kumo, Maho Yagi-Utsumi, Nicola J. Baxter, Koichi Kato, Mike P. Williamson, and Ryo Kitahara, “Close Identity between Alternatively Folded State N2 of Ubiquitin and the Conformation of the Protein Bound to the Ubiquitin-Activating Enzyme” Biochemistry, 53 (3), pp 447–449 (2014) IF=3.0
79. Junichi Iwakiri, Tomoshi Kameda, Kiyoshi Asai, Michiaki Hamada “Analysis of base-pairing probabilities of RNA molecules involved in protein-RNA interactions” Bioinformatics, 29 (20), 2524-2528 (2013) IF=4.6
80. Kawamura, Shuhei; Unno, Yuka; Tanaka, Motohiro; Sasaki, Takuma; Yamano, Akihito; Hirokawa, Takatsugu; Kameda, Tomoshi; Asai, Akira; Arisawa, Mitsuhiro; Shuto, Satoshi, “Investigation of the Non-Covalent Binding Mode of Covalent Proteasome Inhibitors around the Transition State by Combined Use of Cyclopropylic Strain-Based Conformational Restriction and Computational Modeling” J. Med. Chem., 56 (14), 5829–5842 (2013) IF=5.5
81. Hirano, Atsushi#; Kameda, Tomoshi#; Shinozaki, Daisuke; Arakawa, Tsutomu; Shiraki, Kentaro,”Molecular Dynamics Simulation of the Arginine-Assisted Solubilization of Caffeic Acid: Intervention in the Interaction” J Phys Chem. b 117 (25), 7518–7527 (2013) IF=3.4 #: equally contributed
82. Takayoshi Tsuzuki, Natsumi Sakaguchi, Takashi Kudoh, Satoshi Takano, Masato Uehara, Takashi Murayama, Takashi Sakurai, Minako Hashii, Haruhiro Higashida, Karin Weber, Andreas H. Guse, Tomoshi Kameda, Takatsugu Hirokawa, Yasuhiro Kumaki, Barry V. L. Potter, Hayato Fukuda, Mitsuhiro Arisawa, and Satoshi Shuto, “Design and Synthesis of Cyclic ADP-4-Thioribose as a Stable Equivalent of Cyclic ADP-Ribose, a Ca2+-Mobilizing Second Messenger” Angewandte Chemie Inter Ed. 52, 6633-6637 (2013) IF=11.3
83. Soichiro Kitazawa, Tomoshi Kameda, Maho Yagi-Utsumi, Kenji Sugase, Nicola J. Baxter, Koichi Kato, Michael P. Williamson, and Ryo Kitahara, “Solution Structure of the Q41N Variant of Ubiquitin as a Model for the Alternatively Folded N2 State of Ubiquitin” Biochemistry 52 (11), 1874-1885 (2013) IF=3.4
84. Sayoko Yamamoto, Ying Zhang, Takumi Yamaguchi, Tomoshi Kameda and Koichi Kato, ” Lanthanide-assisted NMR evaluation of a dynamic ensemble of oligosaccharide conformations” Chem Comm 48 (39), 4752–4754 (2012) IF=6.4
85. Keisuke Ikeda, Tomoshi Kameda, Erisa Harada, Hideo Akutsu, and Toshimichi Fujiwara, ”Combined Use of Replica-Exchange Molecular Dynamics and Magic-Angle-Spinning Solid-State NMR Spectral Simulations for Determining the Structure and Orientation of Membrane-Bound Peptide” J Phys Chem b 115 9327-9336 (2011) IF=3.7
86. Terakawa T, Kameda T and Takada S, “On easy implementation of a variant of the replica exchange with solute tempering in GROMACS”J Comp Chem 7 1228-1234 (2011) IF=4.6
87. Hirano A#, Kameda T#, Arakawa T, Shiraki K, ”Arginine-Assisted Solubilization System for Drug Substanced: Solubility Experiment and Simulation”J Phys Chem B 114 13455-13462 (2010) IF=3.6#: equally contributed
88. Li W, Yoshii H, Hori N, Kameda T and Takada S, “Multiscale methods for protein folding simulations” Methods 52 106-114 (2010) IF=3.8
89. Maho Yagi-Utsumi, Tomoshi Kameda, Yoshiki Yamaguchi and Koichi Kato, “NMR characterization of the interactions between lyso-GM1 aqueous micelles and amyloidβ” FEBS Lett. 19;584(4) 831-6 (2010) IF=3.6
90. Koga N.#, Kameda T.#, Okazaki K, Takada S, “Paddling mechanism for the substrate translocation by AAA+ motor revealed by multiscale molecular simulations” Proc. Natl. Acad. Sci. 106 (43) 18237-18242 (2009) IF=9.4 #: equally contributed
91. Kameda T., Takada S, “Secondary structure templates the folding of nearby polypeptides” Proc. Natl. Acad. Sci. 103 (47) 17765-17770 (2006) IF=9.6
92. Kameda,T. “Importance of sequence specificity for predicting protein folding pathways: Perturbed Gaussian chain model” Proteins: Structure, Function, and Genetics 53 616-28 (2003) IF=4.3
<総説(国際誌)>
1.
Atsushi Hirano, Kentaro Shiraki, Tomoshi Kameda. Effects of Arginine on Multimodal
Chromatography: Experiments and Simulations. Curr. Protein. Pept. Sc. (2017) doi:
10.2174/1389203718666171024115407, IF=2.7
<論文(国内)>
1. 渡邉 元樹, 山田 恭正, 来見田 遥一, 亀田 倫史, 助野 真美子, 飯塚 まひろ, 曽和 義広, 飯泉 陽介, 高倉 英樹, 宮本 真吾, 増田 光治, 成田 匠, 酒井 敏行, 武藤 倫弘, ケモプロテオミクスおよび分子動力学シミュレーションが明らかにした、延命草の苦味成分ラブドシアノンIの標的タンパク質と抗腫瘍メカニズム: 電気泳動 65(Suppl.) s67-s67 (2021)
2. 平野 篤, 亀田倫史、溶解剤としてのアルギニン:難水溶性薬剤への応用:生物物理 54 (1) (2014)
3. 亀田倫史, シミュレーションで探るアミロイドの世界:生物工学会誌, 84 (10) 407-409(2006)
4. 亀田倫史, Generalized Born エネルギーの妥当性:レプリカ交換分子動力学法によるサンプリング, 物性研究, 86-1(2006)
<雑誌記事など>
1)
亀田倫史, レプリカ交換MD法,
生物工学会誌, 84 (2) 72(2006)
2)
亀田倫史, 簡便に構造サンプリングの幅を広げるレプリカ交換法,
Bionics 8 23(2005)
<招待講演・依頼講演>
1.
分子動力学シミュレーションと機械学習を用いたペプチド凝集傾向予測、蛋白質科学会(web)2021、6月
2.
分子動力学シミュレーションと機械学習を用いたペプチド凝集傾向予測、CBI学会講演会(web)2021、5月
3.
人工知能・分子シミュレーションを用いたタンパク質高機能化法、酵素工学研究会(web)2021、4月
4.
タンパク質生産性を向上させる遺伝子配列設計技術、日本農芸化学会(web)2021、3月
5.
分子動力学シミュレーション機械学習に基づく蛋白質高機能化、日本化学会(web)2021、3月
6.
人工知能・MDを用いた蛋白質高機能化、新化学技術推進協会(web)2020、12月
7.
遺伝子配列設計による蛋白質高発現化・高機能化、BioJapan2020(横浜)2020、10月
8.
Optimizing Gene Sequences for Improved Protein Expression
in Industrial Microorganisms, Peptalk(サンディエゴ、アメリカ)2020、1月
9.
PDBj活用例:MDシミュレーション、トーゴ―の日シンポジウム(東京)2019、10月
10. current trend of
phase separation、第57回日本生物物理学会(宮崎)2019、9月
11. 人工知能・分子シミュレーションを活用した酵素高機能化、第408回CBI学会講演会(東京)2019、7月
12. 天然変性タンパク質p53を標的としたペプチドの人工設計-液液相分離の制御-、第19回日本蛋白質科学会(神戸)2019、6月
13. 人工知能・分子シミュレーションを用いた蛋白質高機能化法、日本生物工学会第6回SBJシンポジウム(大阪)2019、5月
14. 亀田倫史、遺伝子配列設計法、第8回合成生物工学シンポジウム、神戸、2018年9月26日
15. 亀田倫史、池部仁善、分子動力学シミュレーションを用いたタンパク質高機能化法の開発、日本生物工学会、大阪、2018年9月7日
16. 亀田倫史、人工知能を用いたタンパク質高機能化、日本生物工学会、大阪、2018年9月7日
17. 亀田倫史、MDシミュレーションを用いた機能性タンパク質の高機能化法の開発、日本農芸化学会2018年度大会、名古屋、2018年3月
18. 12. 亀田倫史, 分子動力学シミュレーションを活用した機能分子設計・創薬支援、第13回理研「デジタルXバイオものづくり」シンポジウム、埼玉、2018年3月
19. 亀田倫史,分子シミュレーションを活用した創薬、2017BIOVIA Forum、東京,2017年6月
20. 亀田倫史,蛋白質・RNA複合体の立体構造予測、第17回日本蛋白質科学会年会,仙台,2017年6月
21. 亀田倫史,分子動力学シミュレーションを活用した創薬支援、生物物理学会東北支部会,仙台,2015年12月
22. 亀田倫史,蛋白質・RNA複合体立体構造予測、第53回生物物理学会年会,金沢,2015年9月
23. 亀田倫史,溶解剤・分離用カラム溶出剤の開発、 JBAセミナー "未来へのバイオ技術"勉強会「数理モデルを産業活性化と健康長寿社会実現に直結させる (2) 物質・エネルギー生産,東京,2014年12月
24. 亀田倫史,分子動力学研究を活用した創薬支援、JBAセミナー "未来へのバイオ技術"勉強会「数理モデルを産業活性化と健康長寿社会実現に直結させる (1)再生医療・創薬支援」,東京,2014年7月
25. 亀田倫史,MDシミュレーションによるアミロイド形成過程の研究、第14回日本蛋白質科学会年会,横浜,2014年6月
26. 亀田倫史,分子動力学シミュレーションを活用した創薬支援、日本薬学会北陸支部特別講演会,富山,2014年1月
27. 亀田倫史,動的性質を生かした創薬研究、第346回CBI学会研究講演会「蛋白質のシミュレーションによる機能の解明」,東京,2013年2月
28. 亀田倫史,分子動力学シミュレーション技術による化合物可溶化メカニズムの解明、第323回CBI学会研究講演会「創薬における物性評価と計算化学」,東京,2012年1月
29. 亀田倫史,蛋白質異常凝集の原理と制御、大阪大学蛋白質研究所・蛋白研セミナー,大阪,2011年4月27-28日亀田倫史,NMRと分子動力学法との融合
30. 亀田倫史,分子動力学法とNMRの融合、第12回若手NMR研究会,滋賀,2011年6月
31. 亀田倫史,創薬分野におけるコンピューターシミュレーションの活用、IBM科学技術計算ソリューションセミナー,神奈川,2010年10月
32. 亀田倫史,NMR構造決定に最小限必要な距離情報を、Gaussian Chain Modelで導出する、大阪大学蛋白質研究所・蛋白研セミナー,大阪,2009年7月
33. 亀田倫史,難溶物質・アミロイドの溶解シミュレーション、第9回日本蛋白質科学会年会,熊本,2009年5月
34. 亀田倫史,新レプリカ交換法REST による,蛋白質変性構造アンサンブルの解明、第8回日本蛋白質科学会年会,東京,2008年6月
35. 亀田倫史,MDによる構造転移の可視化、第45回日本生物物理学会年会,横浜,2007年12月
36. 亀田倫史,蛋白質シミュレーションにおける水分子モデルの影響、第7回日本蛋白質科学会年会,仙台,2007年5月
<企画・オーガナイザー>
1.
後藤 祐児、宗 正智、白木 賢太郎、亀田倫史,第3回LLPS研究会(蛋白研セミナー:液-液相分離の新たな展開へ向けて),大阪,2019年9月
2.
亀田倫史,第2回LLPS研究会,東京,2019年3月
3.
亀田倫史,第1回LLPS研究会,東京,2018年8月
4.
鎌形清人,亀田倫史,蛋白質・核酸共存系に対する実験家と理論家の挑戦、第53回生物物理学会年会,金沢,2015年9月
5.
藤原敏道,児嶋長次郎,亀田倫史,実験と計算機科学で解明する蛋白質機能構造、大阪大学蛋白質研究所・蛋白研セミナー,大阪,2009年7月
6.
亀田倫史,白木賢太郎,凝集を考える理論と実験、第9回日本蛋白質科学会年会,熊本,2009年5月
<報道>
1.
酵素設計 活性5倍 AIで開発効率化、日刊工業新聞 2021.12.16
2.
産総研ほか 酵素機能の改良作業が計算科学で数百倍向上、日刊ケミカルニュース(WEB)2021.10.21
3.
産総研など、酵素の改変部位を予測する計算法開発 目的化合物の生産6.4倍、日刊工業新聞 2021.10.06
4.
酵素機能改良を効率化 産総研-神戸天然物化学 計算科学の新手法、化学工業日報2021.10.05
5.
計算科学による酵素機能改良の効率化-酵素改変部位の特定により目的の化合物の割合が向上-、JPubb(WEB)2021.10.04
6.
ラブドシアノンI、大腸がん細胞で増殖抑制の分子機序解明-京都府医大ほか、医療NEWS(WEB)、2021.03.15
7.
生薬の苦味成分が大腸がん細胞の増殖を抑制することを府立医大などが確認、マイナビニュース(WEB) 2021.03.10
8.
医薬品候補ペプチド設計 天然変性領域を標的に、 薬事日報,2019.7.29
9.
蛋白質をAIで自動設計、日本工業新聞、2018.9.6
10. たんぱく質の機能改変 AI使い大幅効率化、 化学工業日報,2018.9.3
11. 人工知能でタンパク質を自動設計-様々な機能性タンパク質開発の加速に期待-、2018.08.31、JPubb(WEB)
12. 不要なタンパク質“オール”で引き込む、京都新聞,2009.10.18
<講義>
1.
亀田倫史,キャリアセミナー、立命館大学,滋賀,2021年11月
2.
亀田倫史,キャリアセミナー、立命館大学,滋賀,2020年10月
3.
亀田倫史,キャリアセミナー、立命館大学,滋賀,2019年10月
4.
亀田倫史,分子動力学シミュレーションを活用した創薬支援、東京大学,東京,2013年11月
5.
亀田倫史,分子動力学法の新たな展開、お茶の水女子大学,東京,2011年11月
6.
亀田倫史,分子動力学計算、産総研・生命情報工学研究センター,東京,2010年11月
7.
亀田倫史,分子動力学シミュレーション、お茶の水大学,東京,2010年8月
8.
亀田倫史,分子動力学シミュレーション、富山県立大学,富山,2007年8月