カルモジュリンのコンフォーメーションの研究 (1998-2002)
Komeiji, Y., Ueno, Y., Uebayasi, M. (2002) FEBS Lett.,
521, 133-139.
Molecular dynamics simulations revealed Ca2+-dependent
conformational change of Calmodulin.
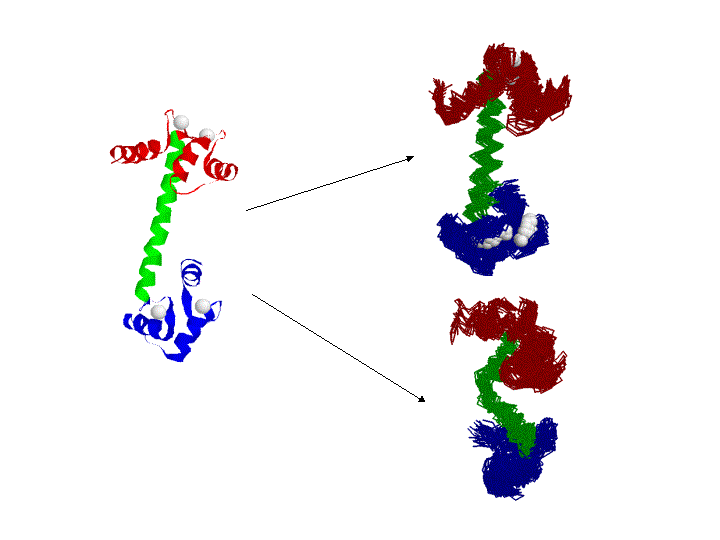
カルモジュリンの結晶構造(左)を出発点にして、 カルシウムイオンがくっついたままの形と(右上)、 除いた形で(右下)MDをしたら、 大きくコンフォーメーションに違いが出た、 というお話です。
細かい部分構造はNMRなどの実験と一致しない部分がありますが、 大局的には、真実の一部を再現しているのでは、 と思っています。つまり、カルシウムがないと、 分子の柔軟性が増し、また構造が縮んで丸くなる。
特におもしろいのは、カルシウムがなくなると、 カルシウムと直接くっついてるわけでもないのに、 中央のヘリックスが曲がってしまうことで、これは、 「イオンの有無によるアロステリック変化をMDで示した」 珍しい例である、と、著者は一人で喜んでますが......?カルモジュリンは、実験屋だった頃に近くに大家がいたり、 似たタンパクのトロポニンを自分で実験で扱ったこともあったので、 随分前から、いろんなMDをやってはみたのですが、 この手のマルチドメイン、 マルチコンフォーメーションのタンパク質は難しい。 やっと論文になった、という感じ。
因みに、PEACH-GRAPEシステムを使っていますが、 ソフトPEACHはともかく、ハードGRAPEを使った研究は、 これが最後でしょう。
カイコガ性フェロモン結合タンパク質のMDシミュレーション (2001-2002)
Nemoto, T., Uebayasi, M., Komeiji, Y. (2002)
CBI J. 2, 32-37.
Flexibility of a loop in a pheromone binding protein from Bombyx mori:
a molecular dynamics simulation.
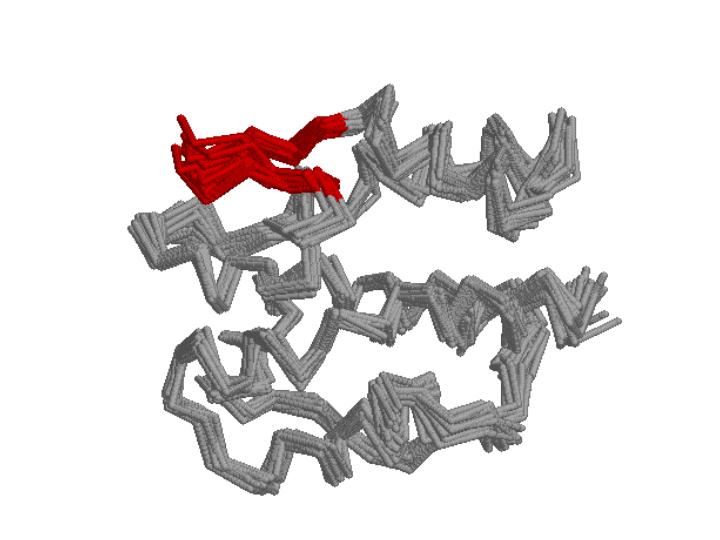
蚕の性フェロモン結合タンパク質に対して、PEACH-GRAPEシステムを使ってMDを行い、 図の赤いループ部分のゆらぎが大きいことを示しました。 で、話をはしょりますが、この部分がフェロモン結合サイトの 「蓋」の役割をしている可能性が高いことを支持する結果なのだそうです。
HIN リコンビナーゼ:DNA複合体の分子動力学法 による研究(1997-1999)
DNA結合タンパク質のMDとしては、 トリプトファン・リプレッサーを対象に行って来たのですが、 この系はわたしの手を離れて別のところでMDが続行中ですし、 もう少し小さくてハンディな系で、DNA結合のタンパク質への効果を 系統的に調べたいと思い選んだのが、この分子。 HIN リコンビナーゼのC末の52残基とDNAとの複合体で、かなり小さく、 またZnフィンガーなどと違ってDNA結合にイオンが直接絡んではおらず、 MDの対象として適切だと思います。 複合体、ペプチド、DNAのそれぞれについて独立にMDを行い、 構造やダイナミクスを比較する、という攻め方で研究しています。
核酸は負に帯電しているため、 クーロン力のノーカットオフ計算が 絶対必要なため、 PEACH-GRAPEシステム を使うメリットが大きいと思われます。
Komeiji, Y., Uebayasi, M. (1999) Mol. Simul. 21, 303-324
Molecular Dynamics Simulation of the Hin-Recombinase-DNA Complex.
まずは複合体のみ、エワルド法で1ナノ秒MDを行った結果を出してみました。 ペプチドの両末端が複合体形成に重要であることなどが示唆 されました。他にエワルド法のパラメータの決定法や、 カットオフMDとの結果比較など。
Komeiji, Y., Uebayasi, M. (1999) Biophys. J. 77, 123-138
Change in conformation by DNA-peptide association:
Molecular dynamics of the Hin-recombinase-hixL complex.
続報で、複合体に加えて、ペプチドとDNAそれぞれの単体のMDも行い、 複合体のダイナミクスと比較したもの。分子認識がinduced fitであること、 分子間の水素結合は、決して静的なものではなく、 時々刻々動的に変化していること、 などを示しました。
電圧依存性イオンチャネルのイオン透過のシミュレーション(1995-1998)
Suenaga, A., Komeiji, Y., Uebayasi, M., Meguro, T., Saito, M.,
Yamato, I. (1998) Biosci. Rep. 18, 39-48.
Computational observation of an ion permeation through a channel
protein.
イオンチャネルタンパク質の中を, イオンが電圧依存で透過する現象を、 世界で初めてMDシミュレーションした論文。計算対象は、OmpFポリンで、 透過イオンはNa+。改造型AMBERを使用。 この論文は、末永氏と山登先生の労作で、古明地は横から口を出していただけです。
生体分子シミュレーションソフトPEACHの開発(古典MD編) (1993-2007)
PEACH (Program for Energetic Analysis of bioCHemical molecules) は、専用計算機GRAPE対応ソフトとして 開発されました(Ver. 1, 1995)。 しかし、 いつまでも、専用計算機ばかりというわけには行かないもんで、 Ver. 2 (1997)では普通の汎用UNIXワークステーションでも 利用できるようになり、さらに、Ver. 3 (2000)では MPIによる並列化もなされています。 Ver. 4 (2003)からは、(F)MO-MDも導入しました。Komeiji, Y. (2000) J. Mol. Struct.: THEOCHEM, 530, 237-243.
ここでは、PEACHのアルゴリズムや利用法に関する論文や、 総説をまとめておきます。
と言いつつ、PEACHの公開もやめてしまったことだし、 手元でちょぼちょぼ改良してはいますが、 とりあえず中締めにしました。なお、古典MDではなく、FMO-MDへのPEACHの利用は、 FMOの項をご覧ください。
Ewald summation and multiple time step methods for molecular dynamics simulation of biological molecules.
PEACHに導入した多重時間刻み幅法と エワルド法を組み合わせる条件について述べた論文です。Komeiji, Y., Uebayasi, M., Nagashima, U. (2000) J. Chem. Software 6, 1-36.
REVIEW: Molecular Dynamics Simulation of Biological Molecules (1) Methods
生体分子の分子動力学シミュレーション(1)方法
Komeiji, Y., Tajima, S., Haraguchi, M., Takahashi, N.,
Uebayasi, M., Nagashima, U. (2001) J. Chem. Software,
7, 1-28.
REVIEW: Molecular Dynamics Simulation of Biological Molecules
(2) Practice.
生体分子の分子動力学シミュレーション(2)応用
続き物の日本語の総説です。PEACHに導入したアルゴリズムを中心に、 生体分子の分子動力学シミュレーションの方法を概観し、さらに、 PEACHを使って生体分子のMDを行う手順を、 実践的に解説しました。"Technical Detail"の嵐です。 MDの初心者で、つまづいている方の役に立つかも知れません (PEACH以外のソフトを使う場合でも)。Komeiji, Y., Haraguchi, M., Nagashima, U. (2001) Parallel Computing, 27, 977-987.
Parallel molecular dynamics simulation of a protein.
PEACH 3.0以降は、第二著者の原口氏がMPIで書き換えてくださったため、 汎用計算機でも並列計算ができます。 この論文は、並列化アルゴリズムの説明とベンチマーク、 GRAPEとの比較などを載せてあります。
専用計算機を用いた生体高分子高速 シミュレーションシステムの構築(1993-2002)
MD-GRAPE という非共有結合力だけを高速に計算する特殊プロセッサと、 PEACHという自作のMDプログラムを用いて 生体分子の高速MDシステム(PEACH-GRAPEシステム)を構築した、 という話です。
Komeiji, Y., Yokoyama, H., Uebayasi, M. Taiji, M. Fukushige, T.,
Sugimoto, D., Takata, R., Shimizu, A., Itsukashi, K. (1995)
In "Pacific Symposium on Biocomputing '96 (Hunter & Klein eds.,
World Scientific)," pp. 472-487.
A High performance system for molecular dynamics simulation of
biomolecules using a special-purpose computer.
a molecular dynamics simulation.
ハワイでのとある国際学会のプロシーディングスです。 PEACHとMD-GRAPEの概要と、簡単な性能評価が載ってます。
Komeiji, Y., Uebayasi, M., Takata, R., Shimizu, A., Itsukashi, K,
Taiji, M. (1997) J. Comput. Chem. 18, 1546-1563.
Fast and accurate molecular dynamics simulation of a protein using a
special purpose computer.
この論文は、PEACH-GRAPEシステム、特にPEACHのドキュメントのような ものなので、長い割にはオリジナルな情報はあまりありません (自分で言っててどうする! )。GRAPEが扱える力や境界条件、 力の計算方法 とソフトウェアインターフェース、PEACHに導入した多時間刻み幅法や定温 アルゴリズムの説明、PEACH-GRAPEシステムによるタンパク質 (ヒスチジン含有タンパク質、HPr)の様々な条件でのMDの結果と、 お約束の従来のカットオフ法との比較、など が載ってます。Komeiji, Y., Uebayasi, M. (2002) CBI J. 2, 102-118.
ここで利用しているエワルド和や直接和によるノーカット計算とRESPAによる 多時間刻み幅法は、1991年の論文(2)で使ったTwin-range法の改良 版とも言えるわけでして、7年かけて古明地はその程度の進歩しか しなかったのでした(そんなもんです)...
Peach-Grape system - a high performance simulator for biomolecules.
とにもかくにも、PEACHのGRAPE対応機能は、Ver.3.0A (2000)を最後に打ち切り、 カルモジュリンの論文で全てに片がついたので、 この総説を書いて、Peach-Grapeシステムの件は、おしまいにしました。 MD専用計算機による生体分子シミュレーション一覧、 専用計算機を有効に用いるためのポイント、など、 割とおもしろいことを書いたつもり。。
アルファヘリックスの安定性に関する 自由エネルギー計算(1992-93)
Komeiji, Y., Honda, N., Yamato, I. (1993) Biophys. Chem. 47, 113-121.Helix propensity of Ala and Val: A free energy perturbation study.
短いぺプチドは、系が小さいからMDやFEPの練習用には、 うってつけですが、小さくて本質的なぶん、却って大きなタンパク質よりも むづかしい面もあります。激しくコンフォーメーションが変化しますから。 この論文は、アルファヘリックスの真ん中のアミノ 酸は、ValよりもAlaのほうが安定性が高い、というのをFEPで再現したもの。 展開すればおもしろいテーマなのですが、これ一発で終わってしまいました。
トリプトファン・リプレッサータンパク質の 分子動力学法による研究(1990-2000)
トリプトファン・リプレッサー:DNA複合体
元来怠け者のわたしは、体力と忍耐力勝負の実験生化学はおちこぼれ、 分子動力学法(MD) による計算機シミュレーションという、生物学の中では河原者の分野に 走ったのでした。トリプトファン・リプレッサーというタンパク質は(以下TrpR)、 大腸菌の体内で、トリプトファンというアミノ酸が多すぎたり少なすぎたり しないように調節しているタンパク質です。
トリプトファンが多くなるー>という図式。
トリプトファンをTrpRがひっつけて形が変わるー>
DNAにひっついて、トリプトファン合成系の転写を阻害ー>
トリプトファンが少なくなる
生物学的にいろいろな相互作用して おもしろいし、いろいろな形の結晶構造もわかっているし(トリプトファン結合/ 非結合、DNA 結合/非結合)、ということで選んだのですが、DNAがくっついて ない形でも分子量2万5千でして、 1990年当時のMDの対象としては巨大でした(1998年の基準でも、決して 小さくはありません)。 以下の研究では、MD計算は、AMBER というカリフォルニア大のKollman groupで開発されたソフトを用いて 行いました。東京理科大の山登先生、生命研の上林さんとの共同研究です。
Komeiji, Y., Uebayasi, M. Someya, J., Yamato, I. (1991) Protein Engng. 4,
871-875.
Molecular dynamics simulation of trp-aporepressor in a solvent.
DNA もトリプトファンもひっついてない形のTrpRを安定にMDを走らす条件 について考察したもの。結局、
(A)水分子を露に取り入れなきゃだめ
(B) クーロン力は一般に使われていた9A程度のカットオフではみじかすぎ、 Twin-range 法で18 A程度まで考慮して、やっと安定になる、
というのが結論。(A)(B)を示すグラフ一枚のおかげで、時々引用されました。
なお、Twin-range法とは、遠くのクーロン力の寄与を 数回に一回計算して補正する方法、この 方法を導入するために、AMBER をかなり書き換えました。方法としては、 古明地のオリジナルではありません。一番最初に誰が考え出したかは 不明です。うわさでは、1960台に物性物理の分野で使われたのが最初で、 それを、Van Gunsterenたちが、生体分子に応用したのだそうです。
Komeiji, Y., Uebayasi, M. Someya, J., Yamato, I. (1992) Protein Engng. 5,
759-767.
Free energy perturbation study on a Trp-binding mutant (Ser88->Cys) of
the trp-repressor.
これは、自由エネルギー摂動法 (以下FEP)という方法で、アミノ酸置換のタンパク質の活性に及ぼす影響を 調べた計算です。ここでは、TrpRのトリプトファン結合部位のSer88をCys に置換した場合の結合活性の変化を計算し文献から取った実験値と比較しま した。実験値と計算値は大体一致して、結合活性が低下するということで それはまあそれでいいのですが、この論文では、結合活性の低下の原因を、 自由エネルギーを分割して、詳しく議論しているのが特徴です(ここでも、 AMBERを書き直して分割法を導入。AMBERの現行バージョン - 4.1や5.0 - は、Twin-range も自由エネルギー分割法も入っているようです)。この 自由エネルギー分割法というのが、あとで議論の対象になりました。
Komeiji, Y., Uebayasi, M. Someya, J., Yamato, I. (1993) Proteins 16,
268-277.
A molecular dynamics study of solvent behavior around a protein.
タンパク質部分の構造やダイナミクスを解析 したMDのトラジェクトリを使って、タンパク質周囲の水 の挙動を解析したものです。 学位を取るための論文稼ぎという説もありますが、 似たような研究をする人が多いせいか、別刷も早目に売り切れたし、 いろんな人が引用してくれます。 わたしの論文の中では最高の被引用数なのですが、 単に他の論文が引用されなさすぎる、といったほうが正しいかも。
Komeiji, Y., Fujita, I., Honda, N., Tsutsui, M., Tamura, T., Yamato, I.
(1994) Protein Engng.7, 1239-1247.
Glycine-85 of the trp-Repressor of E. coli is important in forming the
hydrophobic tryptophan binding pocket.
FEPの延長で、今度は計算と実験を実際に一緒にやって比較してみた 論文です。トリプトファン結合部位のGly85をいろいろなアミノ酸に変えて みました。実験は、当時理科大の院生だった藤田いづみさん、計算の一部は 本多信雄君に担当してもらいました。実験値と計算値は定性的には一致しま した。しかし、レフェリーに、「計算値の誤差が実験値の誤差より大きいか らだめだ」とか言われたり、自由エネルギーの分割 論争に巻き込まれたりして、計算の部分の記述はだいぶ削らざるを得ません でした。
Komeiji, Y., Uebayasi, M., Yamato, I. (1994) Proteins 30, 248-258.
Molecular dynamics simulations of the trp apo- and holo-repressors:
domain structure and ligand-protein interaction.
TrpRにトリプトファンがついた形とついてない形でそれぞれ MDを行い、ダイナミクスを比較した論文です。まあ、タンパク質部分に ついては、今回調べた限り、あんまりダイナミクスやらなにやらの差はみられ なかったのですけどねえ.....むしろ、基質のトリプトファンとタンパク 部分の相互作用についてのほうが、クリアな結果がでました。TrpRに関して、古明地が主体的に行った分はこの論文でおしまい。 残りの論文は、理科大の学生さんたちのデータばかりです。
Fujita, I., Komeiji, Y., Yamato, I. (1995) Protein Engng. 8, 935-938.
Threonine 81 of the trp-repressor of E. coli plays an auxiliary role
for the formation of the corepressor binding pocket"
Thr81の変異についても実験と計算を比較しようとしました。 しかし、実験だと、変異による活性変化は小さく、せいぜい1kcal/mol程度。 これは、当時われわれがやっていたFEP計算では、完全に誤差の範囲で、 とても世に出せるような計算結果が出ませんでした。結局、藤田さんの実験 データだけを論文にしました。
Honda, N., Komeiji, Y., Uebayasi, M., Yamato, I. (1996) Proteins 26,
459-464.
Computational design of a substrate specificity mutant of a protein.
自由エネルギーの分割というのは、たとえば 結合活性の変化は、タンパク質のどのアミノ酸の相互作用がきいているか、と いうような情報を与えてくれる方法です。詳しくは省きますが、本多君がこの 情報を駆使して計算で結合活性を人為的に設計し、実験で設計どおりの タンパク質ができているのを確かめました。そのデータを論文にしようとした 矢先、Van GunsterenのグループからSuenaga, A., Yatsu, C., Komeiji, Y., Uebayasi, M., Meguro, T., Yamato, I. (2000) J. Mol. Struct. 526, 209-218.
「自由エネルギーの分割は、相関のある系 では意味がない」
という議論が出され(Mark & Gunsteren, 1994, J. Mol. Biol. 240, 167)、そのあおりを食って 論文がそのままでは出せなくなりました。こちらとしては、分子設計に 使えるのだから、無意味なはずがないと思ったのですが、なかなか理論的に 反駁できません。でも、結局、
「全体を一様に変化させるなら、相関項が平等 に分配されるから意味がある」
というKarplusたちの反論が出たので (Boresch & Karplus, 1995, J. Mol. Biol., 254, 801)、その尻馬に乗って なんとかこの論文を世に出すことができました。印刷されたのは本多君の データが出てから約3年後のことです。
Molecular dynamics simulation of trp-repressor/operator complex: analysis of hydrogen bond patterns of protein-DNA interaction.
随分時間が空いていますが、TrpR/DNA複合体のMDで、 水を介した水素結合の解析など。TrpR関係は、すべて、 AMBERを使っているのですが、これのみ、 PEACH-GRAPEシステムを使用しています。
TrpRに関する古明地のcontributionは、 これで打ち止めです。さよ~なら~....
プロリン輸送タンパク質の生化学的研究(1987-89)
Komeiji, Y., Hanada, K., Yamato, I., Anraku, Y. (1989) FEBS Lett. 256,
135-138.
Orientation of the carboxyl teminus of the Na+/proline symport
carrier in Escherichia coli.
これは、わたしが真面目に実験生化学をしていた修士のときのデータを発表した もので、古明地がかつては実験も出来た、ことの唯一の証明となって おります。 プロリン輸送タンパク質(PutP)は、大腸菌の内膜で、ナトリウムイオンの電気化学 ポテンシャル差を利用して、濃度差に逆らってプロリンを膜の内側に輸送する タンパク質です。 もっとも、わたしがやっていた実験は、このタンパク質のC末端が膜のどちら側に 出ているか、という渋めのものでしたが。結論は 「細胞質側」 でした。ところで、9年後、C末だけでなく、PutP全体のトポロジーを調べた論文が 出ました(Jung et al., 1998, J. Biol. Chem. 273: 26400-)。 こういうのは、 若気の至りを後になって蒸し返されるようで嫌なもんですが、 幸いにも、「C末が細胞質側」というのは、 彼らの論文でも再現されたようです (ホっ)。
その他の論文
Suenaga, A., Komeiji, Y., Uebayasi, M., Meguro, T., Yamato, I. (1998) J. Chem. Software 4, 127-142. Molecular dynamics simulation of unfolding of histidine-containing phosphocarrier protein in water.
Komeiji, Y., Harata, K., Ueno, Y., Uebayasi, M. (2000)
JCPE J. 12, 39-48.
Anisotropic motion within a protein: comparison between
X-ray crystallography and molecular dynamics simulation
of Human Lysozyme.
VanSchouwen, B. M. B., Gordon, H. L., Rothstein, S. M., Komeiji, Y.,
Fukuzawa, K., Tanaka, S.
Nakano, T. (2008)
Comput. Biol. Chem. 32, 149-158.
Water-Mediated Interactions in the CRP-cAMP-DNA Complex:
Does water mediate sequence-specific binding at the DNA primary-kink site?
Hanyu, Y., Komeiji, Y., Kato, M. (2019) Molecules 24, 2949. Potentiating Antigen-Specific Antibody Production with Peptides Obtained from In Silico Screening for High-Affinity Against MHC-II.
Nakamura, J., Maruyama, Y., Tajima, G., Komeiji, Y., Suwa, M., Sato, C. (2021). Int. J. Mol. Sci. 22, 2624. Ca2+-ATPase molecules as a calcium-sensitive memebrane-endoskeleton of sarcoplasmic reticulum.
Tanaka, S., Komeiji, Y. (2022). J. Phys. Chem. Res. 4, 1-2. Thermal and quantum fluctuations of harmonic oscillator.[full text] [full text in arXiv]